工学院陈匡时课题组发表单分子DNA成像技术的综述
2019/02/18 信息来源: 工学院
编辑:白杨 |工学院生物医学工程系陈匡时课题组在《基因组蛋白质组与生物信息学报》(Genomics Proteomics & Bioinformatics,IF = 6.6)发表综述文章"Progress and Challenges for Live-cell Imaging of Genomic Loci Using CRISPR-based Platforms",介绍了DNA单分子成像技术的现阶段研究进展及面临的挑战,其中重点介绍了基于CRISPR基因编辑技术的成像体系。
Cas9内切酶与single guide RNA (sgRNA)是CRISPR基因编辑系统的重要组成部分。Cas9与sgRNA结合后所形成的复合体可以在sgRNA介导下识别与结合特定DNA序列,并切割目的DNA,通过后续的DNA修复过程造成序列改变,从而达到定点编辑基因组的目的。研究者们发现,失去酶切活性的Cas9 (nuclease-deactivated Cas9, dCas9)仍具备结合sgRNA靶向特定DNA序列的能力,这一发现推动了CRISPR 成像技术的发展。本综述分别阐述了基于荧光蛋白(FP)(图1 A)、有机荧光染料(图1B)以及量子点(QD)(图1C)等不同发光元素的CRISPR成像体系的工作原理,介绍了现阶段每个体系在活细胞标记特定染色质位点、观测其时程变化并获取其动力学信息等方面的应用,最后讨论了CRISPR成像技术所面临的挑战以及可能的解决方案。综述中也介绍了本课题组设计的基于有机荧光染料的CRISPR成像体系—CRISPR/分子信标(CRISPR/MB)的工作(文章链接: https://doi.org/10.1093/nar/gky304)。综述或能帮助研究者更加深入地阐述染色质的构象变化与动态行为,进而为疾病发生与发展的相关研究提供有价值的信息。
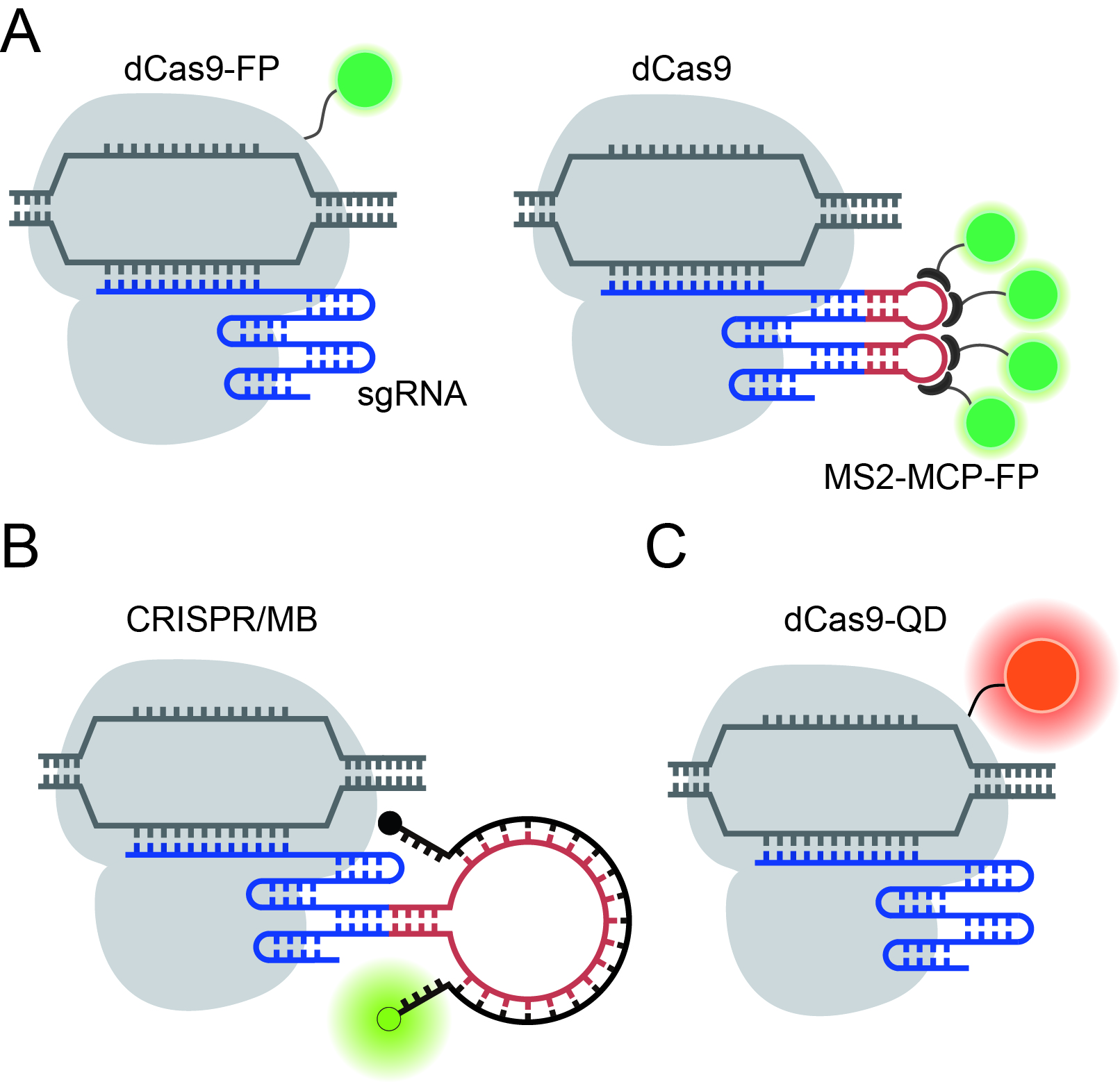
图1. 基于荧光蛋白(A)、有机荧光染料(B) 以及量子点(C) 的CRISPR 成像技术代表
前沿交叉学科研究院博士生吴小天和工学院博士生毛诗琦是该论文的共同第一作者,工学院本科生应亚宸以及北京大学/佐治亚理工/埃默里大学生物医学工程联合博士培养项目的Christopher J. Krueger为工作的顺利完成作出重要贡献。通讯作者为工学院陈匡时特聘研究员。该工作得到了国家重点研发计划、国家自然科学基金、北京市自然科学基金的支持。
转载本网文章请注明出处